designed for easy size determination and DNA quantification
UltraScript® Reverse Transcriptase & cDNA Synthesis Kits UltraScript® 逆転写酵素は、cDNA 合成速度と収量を向上させ、正確な転写表現を実現するように設計された、堅牢で耐熱性に優れた改良型 MMLV 逆転写酵素です。
この酵素はウイルス RNA 配列の高感度逆転写を可能にするため、RT-qPCR ベースの検出テストの理想的なコンポーネントとなります。スタンドアロン酵素として、または逆転写酵素、バッファー システム、およびオプションでアンカー オリゴ (dT) プライマーを含むランダム ヘキサマーの最適化されたブレンドを含むレディ ミックスとしてご利用いただけます。この逆転写酵素は、幅広い RNA テンプレート濃度にわたって、偏りのない、効率的で感度の高い cDNA 合成を提供します。
5’ および 3’ mRNA 転写末端の偏りのない表現
RNase H 活性の低減
耐熱性逆転写酵素 45 °C ~ 55 °C
高度な RNase 阻害剤
わずか 4 pg の総 RNA から高い cDNA 収量を実現
GC リッチ テンプレートの正確な逆転写
低コピー数の転写の高感度検出
Mg および dNTP を含む高度なバッファー化学
プレミックスまたは個別のオリゴ cDNA 合成キットが利用可能
ランダム ヘキサマー、オリゴ (dT)、遺伝子特異的プライマー
リアルタイム PCR 分析用の cDNA 合成
PCR 分析、クローニング、ライブラリ作成、次世代シーケンシング用の cDNA 合成
コピー数が少ない転写産物
ウイルス RNA ターゲット
miRNA ターゲット
全 RNA またはポリ (A)+ RNA からの効率的な合成
UltraScript® Reverse Transcriptase とは? UltraScript® Reverse Transcriptase は、cDNA 合成用の熱安定性が高く非常に活性の高い修飾 MMLV 逆転写酵素 (RTase) です。反応温度は 55 °C まで上げることができ、高い二次構造を持つ GC リッチ RNA 領域のより高い特異性と効率的な転写が可能になります。RTase には、RNase の混入による RNA の分解を防ぐ高度な RNase 阻害剤がブレンドされています。UltraScript® Reverse Transcriptase はリボソーム RNA やトランスファー RNA によって阻害されないため、トータル RNA が理想的な基質になります。
この酵素には、わずか 4 pg の RNA から高収率の cDNA を生成するように最適化された、Mg、dNTP、安定化剤およびエンハンサーを含む 5x バッファーが付属しています。オリゴが含まれていないため、UltraScript® Reverse Transcriptase は、必要な分析の種類に応じてユーザーが独自のプライミング戦略を定義できる利便性と柔軟性を提供します。UltraScript® Reverse Transcriptase は、遺伝子特異的プライマー、オリゴ (dT)、ランダム ヘキサマーを使用して優れたパフォーマンスを発揮し、さまざまな下流アプリケーションに最適な高品質の cDNA を生成します。
PCR Biosytems 社の強力な RTase は、高濃度フォーマットでも、既製の UltraScript® cDNA Synthesis Kit (旧称 qPCRBIO cDNA Synthesis Kit ) としても利用できます。ランダム ヘキサマーとアンカー オリゴ (dT) の相対濃度は、リアルタイム PCR 実験で使用する cDNA の生成用に最適化されています。これにより、プライミング戦略、逆転写酵素、バッファー、エンハンサーなどの重要な逆転写反応因子をユーザーが最適化する必要がなくなり、偏りのない、効率的かつ高感度な cDNA 合成が毎回得られます。
cDNA 合成実験を最大限に活用するために、こちらのヒントとコツ
UltraScript® Reverse Transcriptase の熱安定性は、Sypro Orange 蛍光アッセイを使用して測定しました。タンパク質を Sypro Orange 色素とともにインキュベートし、温度を徐々に上昇させます。タンパク質が展開するにつれて蛍光強度が増し、融点はタンパク質の 50% が展開される温度です。DSF 曲線は、UltraScript® Reverse Transcriptase (紫) と 0.1 mg/ml の野生型 MMLV RT (黒) を示しています。この実験は、UltraScript® Reverse Transcriptase が野生型酵素より 9.3 °C 高い 54.9 ± 0.3 °C で展開することを示しており、この酵素がより熱安定性が高く、反応中に活性を維持する可能性が高いことを示しています。
マウス肝臓から採取した全RNA 100 ngから、UltraScript® Reverse Transcriptase (紫) と競合製品のミックス (青) を使用してcDNAを作成しました。プライマーとして5 μM オリゴdT(18) と5 μM ランダムヘキサマーを加えました。反応物を 42 °C で 10 分間インキュベートしました。得られた cDNA を qPCRBIO SyGreen® Mix を使用して定量しました。PCR Biosystems UltraScript® Reverse Transcriptaseは、同じ時間で競合製品の10倍以上のcDNAを作成しました。
UltraScript® cDNA Synthesis Kit を使用して、マウスの総 RNA を 40 pg から 400 ng まで 10 倍連続希釈して cDNA を合成しました。qPCR は、マウス ACTG 遺伝子の 122 bp フラグメントを増幅する qPCRBIO SyGreen Mix を使用して実行されました。テストした範囲全体で効率は 96% と測定されました。
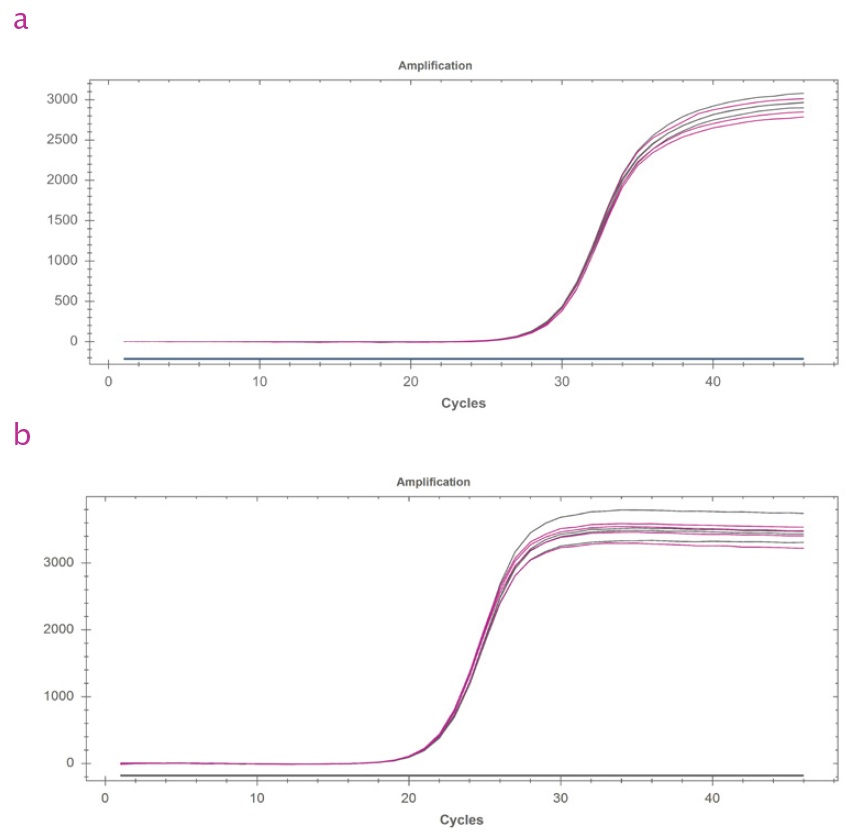
a) UltraScript® cDNA Synthesis Kit を使用して、マウス肝臓の全 RNA から cDNA を合成しました。4.2 kb のマウス CANX 転写産物の 5’ (ピンク色のトレース) および 3’ (灰色のトレース) 末端に対して 2 つのプライマーペアが設計されました。qPCRBIO SyGreen® Mix を使用して分析を行いました。プライマーペアは 4 kb 離れており、逆転写バイアスは示されなかったため、増幅トレースは重複しています。
b) RNS18 遺伝子 (1.8 kb) の 5’ (ピンク) および 3’ (灰色) トレースに対する 2 つのプライマーペア。この場合も、逆転写バイアスは見られませんでした。
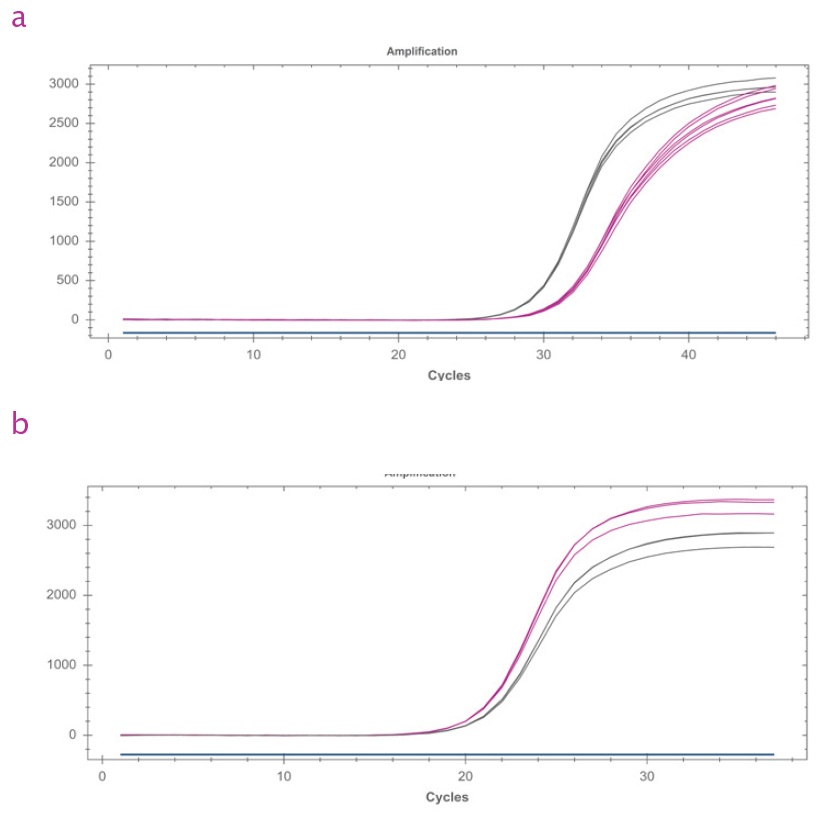
a) UltraScript® cDNA Synthesis Kit を使用して、42 ℃ (ピンク) および 55 ℃ (灰色) でマウス肝臓のトータル RNA から cDNA を合成しました。プライマー ペアは GJB2 に対して設計され、84% GC アンプリコンを生成しました。qPCRBIO SyGreen Mix® を分析に使用しました。高温でのインキュベーションでは、低温でのインキュベーションよりも GC リッチな cDNA が生成されました。
b) GAPDH からの 55% GC のコントロール アンプリコンは、上記の 2 つの cDNA から増幅されました。この GC% では、高温でのインキュベーションによる利点は得られませんでした。温度が高くなると、収量はわずかに低下しました。
UltraScript® cDNA Synthesis Kit
UltraScript® cDNA Synthesis Kit Separate Oligos
UltraScript® Reverse Transcriptase
UltraScript® Reverse Transcriptase (2500 U/μL)
UltraScript® Reverse Transcriptase (8000 U/μL)
長いターゲット (15 ~ 20 kb) を UltraScript® RTase で逆転写できますか?
RTase はプライミングされた基質にジャンプオンしたりジャンプオフしたりするため、当社のすべての RTase で 20 kb の RNA 逆転写を実現できます。二次構造、非特異的結合、およびミスプライミングは、反応が途中で終了する最も一般的な原因となります。
特定のプライマーまたはオリゴ (dT) を使用し、反応の各側面 (たとえば、さまざまな酵素濃度、プライマー、プライマー濃度、温度、温度サイクル、時間など) を最適化することをお勧めします。また、その後の PCR/qPCR ステップの最適化が必要になる場合もあります。
長いテンプレートの逆転写には、より長時間高温に耐えられる UltraScript® 2.0 RTase をお勧めします。これは、RNA の長さに応じて二次構造の可能性が高まるため必要になる場合があります。
参考までに以下の論文を掲載します[1]。
Thiel, V. et al. Effective amplification of 20-kb DNA by reverse transcription PCR. Anal Biochem 252 , 62-70, doi:10.1006/abio.1997.2307 (1997).
UltraScript® cDNA Synthesis Kit を使用して miRNA を増幅できますか?
miRNA は、UltraScript® cDNA Synthesis Kit を使用して増幅しないでください。UltraScript® cDNA Synthesis Kit Separate Oligos 、UltraScript® Reverse Transcriptase 、または UltraScript® 2.0 Reverse Transcriptase を使用する必要があります。これらの製品には、miRNA 固有のプライマーに干渉する可能性のあるランダム ヘキサマーとオリゴ (T) を含まないバッファーが付属しているからです。
UltraScript® Reverse Transcriptase を使用して miRNA を増幅できますか?
当社の RTase はすべて、miRNA の定量と分析に使用できます。ただし、専用のキットは販売しておりません。
次の 2 つのアプローチのいずれかを使用することをお勧めします。
ユニバーサル RT プライマーを使用して、ポリ (A) またはポリ (U) テールを追加し (例: ポリ (U) ポリメラーゼによる)、続いてユニバーサルプライマーを使用して cDNA を合成します[1],[2]。
特定の RT プライマーを使用し、テーリング ステップを省略します[1],[3]-[5]。
これらのアプローチの詳細がよくわからない場合は、ガイドラインとして役立つ以下の参考リストを参照してください。
Dave, V. P. et al. MicroRNA amplification and detection technologies: opportunities and challenges for point of care diagnostics. Lab Invest 99 , 452-469, doi:10.1038/s41374-018-0143-3 (2019).
Mei, Q. et al. A facile and specific assay for quantifying microRNA by an optimized RT-qPCR approach. PLoS One 7 , e46890, doi:10.1371/journal.pone.0046890 (2012).
Chen, C. et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res 33 , e179, doi:10.1093/nar/gni178 (2005).
Raymond, C. K., Roberts, B. S., Garrett-Engele, P., Lim, L. P. & Johnson, J. M. Simple, quantitative primer-extension PCR assay for direct monitoring of microRNAs and short-interfering RNAs. RNA 11 , 1737-1744, doi:10.1261/rna.2148705 (2005).
Androvic, P., Valihrach, L., Elling, J., Sjoback, R. & Kubista, M. Two-tailed RT-qPCR: a novel method for highly accurate miRNA quantification. Nucleic Acids Res 45 , e144, doi:10.1093/nar/gkx588 (2017).
UltraScript® cDNA Synthesis Kit はエンドポイントおよび qPCR に使用できますか?
当社のすべての RTase とキットは、エンドポイントと qPCR アプリケーションの両方に使用できます。
UltraScript® cDNA Synthesis Kit は、真核生物および原核生物の RNA を逆転写できますか??
UltraScript® cDNA Synthesis Kit は、真核生物と原核生物の両方の RNA を増幅できます。当社のミックスには、十分に長いフラグメントが含まれている限り、すべてのタイプの RNA のユニバーサル プライマーであるランダム ヘキサマーが含まれています。ヘキサマーが存在するため、RTase で利用できる場合は、すべての生物の RNA を逆転写できます。このミックスには、真核生物の mRNA などの A テール RNA に作用するオリゴ d(T) も含まれています。
より選択的なキットの場合は、UltraScript® Reverse Transcriptase または UltraScript® 2.0 Reverse Transcriptase を使用してください。これらの製品のバッファーにはヘキサマーやオリゴ (dT) が含まれていません。これにより、独自のプライミング戦略を決定できます。
UltraScript® Reverse Transcriptase は血液または血漿サンプルに使用できますか??
はい、ただし、血液中に存在するさまざまな阻害剤の処理には UltraScript® 2.0 のほうが適しているため、アプリケーションでは UltraScript® 2.0 と比較することをお勧めします。また、RT 反応の血液/血漿サンプルの量を滴定して最適化することもお勧めします。
UltraScript® Reverse Transcriptase はテンプレートの切り替えを行うことができますか?
当社の RTase はすべて、テンプレート切り替えアプリケーションで使用できます。現時点では完全なプロトコルや専用キットは提供していませんが、参考として下記の記事のリストを参照することをお勧めします。
Takada, S. & Mano, H. Profiling of microRNA expression by mRAP. Nat Protoc 2 , 3136-3145, doi:10.1038/nprot.2007.457 (2007).
Picelli, S. et al. Full-length RNA-seq from single cells using Smart-seq2. Nat Protoc 9 , 171-181, doi:10.1038/nprot.2014.006 (2014).
Moldovan, N. et al. Multi-Platform Sequencing Approach Reveals a Novel Transcriptome Profile in Pseudorabies Virus. Front Microbiol 8 , 2708, doi:10.3389/fmicb.2017.02708 (2017).
Zajac, P., Islam, S., Hochgerner, H., Lonnerberg, P. & Linnarsson, S. Base preferences in non-templated nucleotide incorporation by MMLV-derived reverse transcriptases. PLoS One 8 , e85270, doi:10.1371/journal.pone.0085270 (2013).
逆転写を実行するにはサーマルサイクラーが必要ですか??
逆転写は等温プロセスなので、安定した温度を維持できる機器であればどれでも問題ありません。汚染の恐れがあるため、ウォーターバスの使用はお勧めしません。サーマルサイクラーでは蓋が加熱されるため、チューブキャップの結露の問題を回避できるというさらなる利点もあります。
UltraScript® と UltraScript® 2.0 Reverse Transcriptase の主な違いは何ですか?
両製品は野生型 MMLV 逆転写酵素から派生したもので、機能を強化する複数の変異を含んでいます。それぞれが優れた RTase です。UltraScript® 2.0 は 65 °C 以上まで耐熱性が高く、阻害剤に耐性があり、最大 3.5 μg の大量の RNA を逆転写できます。UltraScript® 2.0 は、存在する RNA の量が少ないために感度が必要なアプリケーションにも使用できますが、これには現在の配合を使用してユーザー側で大幅な最適化が必要となるため、代わりに UltraScript® を使用することをお勧めします。UltraScript® は 1 ステップ キットで使用できますが、UltraScript® 2.0 は 2 ステップ プロセス (RT、PCR) の一部としてのみ使用してください。UltraScript® 2.0 は、大量の二次構造を含む長くて扱いにくいテンプレートに特に推奨されます。両方の RTase は、ヘキサマーおよびオリゴ d(T) の有無にかかわらず cDNA 合成キットで使用できます。
UltraScript® Reverse Transcriptase のダイナミック レンジはどのくらいですか?
UltraScript® RTase は、わずか 4 pg から最大 0.4 μg の RNA を転写できます。パフォーマンスは、RNA の種類、RNA の品質、および特定のプライマー、オリゴ (T)、またはランダム ヘキサマーが使用されているかどうかによって異なります。より大容量のアプリケーションには、UltraScript® 2.0 Reverse Transcriptase および cDNA Synthesis Kit を使用してください。
UltraScript® Reverse Transcriptase のエラー率はどのくらいですか??
市販されている大部分の RTase と同様、UltraScript® Reverse Transcriptase および当社が提供する他のすべての RTase は、野生型モロニーマウス白血病ウイルス (MMLV) RTase に由来しており、エラー率は 1×10-4 エラー/bp です[1]。これは、鳥類骨髄芽球症ウイルス (AMV) 由来の RTase にも当てはまります反応を高温で実行すると、ミスマッチの塩基対が不安定になるため、忠実度が高まります[2]。忠実度を低くする必要がある場合は、マンガンを追加すると RTase の変異原性が高まり、速度が速くなる可能性があります[3]。
Yasukawa, K. et al. Next-generation sequencing-based analysis of reverse transcriptase fidelity. Biochem Biophys Res Commun 492 , 147-153, doi:10.1016/j.bbrc.2017.07.169 (2017).
Malboeuf, C. M., Isaacs, S. J., Tran, N. H. & Kim, B. Thermal effects on reverse transcription: improvement of accuracy and processivity in cDNA synthesis. Biotechniques 30 , 1074-1078, 1080, 1082, passim, doi:10.2144/01305rr06 (2001).
Cadwell, R. C. & Joyce, G. F. Randomization of genes by PCR mutagenesis. PCR Methods Appl 2 , 28-33 (1992).
UltraScript® cDNA Synthesis Kit を使用して生成できる生成物の最大長はどれくらいですか?
UltraScript® cDNA Synthesis Kit に含まれる RTase は、十分な最適化を行うことで最大 20 kB の非常に長いターゲットを転写できます[1]。ただし、キットにはランダムヘキサマーが含まれており、配列に沿ってランダムな場所に整列するため、連続生成物の最大長が大幅に短縮されます。ほとんどの生成物は大部分が不連続ですが、RNAの配列全体がカバーされ、qPCRで定量化できます。これは、ランダムヘキサマーを組み込んだcDNA合成キットすべてに当てはまります。完全長の連続生成物が必要な場合は、ヘキサマーやオリゴ(dT)を含まないバッファーが付属するUltraScript® Reverse TranscriptaseまたはUltraScript® 2.0 Reverse Transcriptaseをお勧めします。
Thiel, V. et al. Effective amplification of 20 kb DNA by reverse transcription PCR. Anal Biochem 252 , 62-70, doi:10.1006/abio.1997.2307 (1997).
cDNA 収量が低い場合、どのようなトラブルシューティングがありますか?
低収量が発生する理由は複数考えられます。
RNA の量または完全性が、後続のエンドポイントまたは qPCR ステップに必要な量の生成物を得るのに不十分である可能性があります。RNA の量を増やしてみるか、ゲル電気泳動や RIN 値を使用して RNA の品質を確認してください。RNA の別の精製プロトコールを検討するか、早い段階で RNase 阻害剤を使用してください。当社のすべての RTase には RNase 阻害剤が含まれていますが、一部のアプリケーションでは RT ステップの前に追加する必要がある場合があります。
RNA 調製物には、ヘム、高濃度の NaCl、SDS、チオシアン酸グアニジン、メラニン、カルシウムなどの阻害剤が含まれる場合があります[1]。反応を阻害する可能性のあるすべての物質を除去する別の精製方法を試してください。反応前に RNA を希釈することもできます。これにより理論上の収量は減少しますが、阻害物質も RTase が効率的に機能するのに適したレベルまで希釈されます。これにより、後続のステップに十分な生成物が得られる可能性があります。
一部の RNA は、二次構造や RTase の理想的な基質ではない配列の連続により、本質的に転写が困難です。RTase を添加する前に、温度を上げたり、70°C で 10 分間 RNA 変性/プライマー アニーリングを実行してみます。
RT ステップの後に RNAseH 処理を使用すると、RNA-DNA ヘテロ二本鎖は DNA-DNA 二本鎖よりも安定であるため、特に GC リッチなターゲットの場合、収量が増加します。
配列がまだ十分に表現されていない場合は、特定のプライマーまたはオリゴ (dT) のみを使用した、よりターゲットを絞ったアプローチを使用する必要があるかもしれません。この目的には、ヘキサマーおよびオリゴ (dT) を含まないバッファーが付属する UltraScript® Reverse Transcriptase または UltraScript® 2.0 Reverse Transcriptase をお勧めします。
Schrader, C., Schielke, A., Ellerbroek, L. & Johne, R. PCR inhibitors – occurrence, properties and removal. J Appl Microbiol 113, 1014-1026, doi:10.1111/j.1365-2672.2012.05384.x (2012).
UltraScript® RTase を使用した後にスメアや非特異的産物が見える場合、どのようなトラブルシューティングがありますか?
RT 反応後にスメアや非特異的生成物が現れるのには、複数の理由が考えられます。トラブルシューティングでは次の点を考慮することができます。
PCR/qPCR ステップが逆転写ステップの下流で実行される場合、問題が逆転写ステップと PCR/qPCR ステップのどちらに関連しているかを確認することが重要です。適切なコントロールを使用して PCR/qPCR 反応のトラブルシューティングを行い、上記の問題を引き起こす可能性のあるステップのリストからこれを除外してください。
スメアリングはプライマーオリゴマー化を示している可能性があります。オリゴマー化は、オリゴマーの設計が不適切で、テンプレートの濃度が低い場合に発生する可能性があります。これにより、プライマーダイマーまたはオフターゲット配列の逆転写が発生します。プライマーの再設計、反応温度の上昇、テンプレートの量の増加、プライマー濃度の低下、または反応時間の短縮をお勧めします。これらの考慮事項は、非特異的バンドにも使用できます。
プライマーはターゲット配列に固有ではない可能性があり、再設計が必要になる場合があります。
反復性の高い配列に対するプライマーはスメアリングを引き起こす可能性があるため、プライマーの再設計を考慮する必要がある場合があります。
RNA が劣化している可能性があります。 RNA の量を増やしてみるか、ゲル電気泳動や RIN 値を使用して RNA の品質を確認してください。RNA の別の精製プロトコルを試すか、早い段階で RNase 阻害剤を使用してください。当社のすべての RTase には RNA 阻害剤が含まれていますが、アプリケーションによっては RT ステップの前に追加する必要がある場合があります。
RNA を取り扱うときは、手袋、エアロゾルバリア付き容積式ピペットなどの使用など、標準的な予防措置を考慮してください。
DNA 汚染がないことを確認してください。
RT-qPCR 分析で偽陽性が発生するのはなぜですか?
RNA サンプルが DNA で汚染されている可能性があります。より適切なプロトコルを使用して RNA を再精製するか、熱不安定性 DNase を使用して汚染を取り除くことをお勧めします。