fast, highly specific and ultra-sensitive probe-based real-time RT-PCR
qPCRBIO Probe 1-Step Go qPCRBIO Probe 1-Step Go は、高速で非常に特異的かつ高感度なプローブベースのリアルタイム RT-PCR 用に設計されたユニバーサル プローブ キットです。逆転写酵素技術とバッファー化学の最新開発を使用して、1 つのチューブで効率的な cDNA 合成とリアルタイム PCR を実現します。
このキットは、TaqMan®、Scorpions®、分子ビーコン プローブなど、幅広いプローブ テクノロジーで使用できるように設計されています。
SARS-CoV-2 を含む RNA ウイルスを迅速かつ高感度に検出
早期の Ct 値に対する急速な伸長速度
市場をリードする感度 – 検出限界の向上
多重反応での高い効率
高度な RNase 阻害剤
抗体媒介ホット スタート技術
標準および高速サイクル条件のすべてのリアルタイム PCR プラットフォームとの互換性
新型コロナウイルス感染症 (COVID-19) / SARS-CoV-2 の診断と研究
リアルタイム PCR 診断
シングルプレックスおよびマルチプレックス
極度にコピー数の少ないターゲットの検出
絶対的な定量化
相対的遺伝子発現解析
TaqMan®、Scorpions®、およびモレキュラービーコンプローブ
qPCRBIO Probe 1-Step Go は、TaqMan®、Scorpions®、分子ビーコンプローブなど、幅広いプローブ技術で使用できるように設計されています。キットには 2 倍ミックスが含まれており、mRNA、全 RNA、ウイルス RNA 配列など、あらゆる RNA テンプレートの定量に使用できます。qPCRBIO Probe 1-Step Go は、幅広いテンプレート濃度で迅速かつ正確な結果を提供するように設計されており、SARS-CoV-2 などの RNA ウイルスの検出に最適です。
このキットには、耐熱性があり極めて活性の高い改良型 MMLV 逆転写酵素 (RTase Go) と、RNase の混入による RNA の分解を防ぐ高度な RNase 阻害剤が含まれています。抗体媒介ホット スタート技術により、プライマー ダイマーと非特異的産物の形成が防止され、マルチプレックスで比類のない効率性を持つ、非常に特異的で超高感度のリアルタイム RT-PCR が実現します。ポリメラーゼ技術の最新開発と高度なバッファー化学を組み合わせることで、最適化を最小限に、または最適化なしで市場をリードするパフォーマンスを提供します。
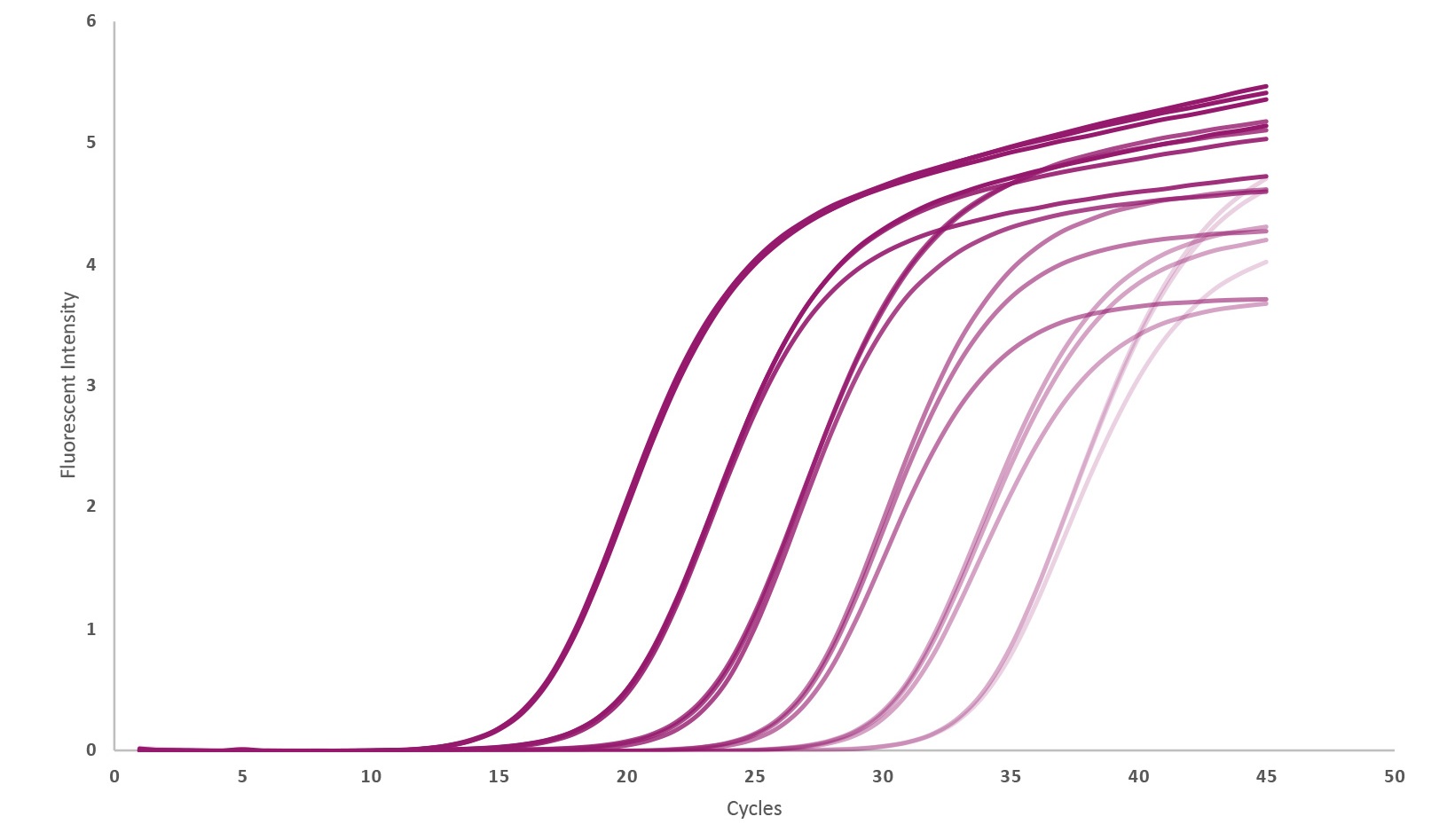
マウス肝臓の全RNAをテンプレートとして3回繰り返して使用し、マウス遺伝子ACTBのTaqMan®プローブ増幅トレースを示します。テンプレート濃度は、20μl反応あたり10pgから1μgの全RNAの範囲の10倍連続希釈です。サイクル条件は、45°C 10分、95°C 3分、その後95°C 10秒、60°C 30秒を45サイクルでした。qPCRBIO Probe 1-Step Goは、幅広いダイナミックレンジにわたって高い効率を示します。
参考データ2:CDC 推奨シーケンスの SARS-CoV-2 N遺伝子の検出 4 つのマウス ハウスキーピング遺伝子が、単一のマルチプレックス反応で同時に増幅されました。テンプレートとして、マウス肝臓全 RNA 1μg を使用しました。増幅は、TaqMan プローブを使用して、次の遺伝子/プローブの組み合わせで検出されました: B2MG/HEX、ACTB/Cy5、GAPDH/FAM、および ACTG/TexasRed
qPCRBIO Probe 1-Step Virus Detect Lo-ROX
qPCRBIO Probe 1-Step Go Hi-ROX
qPCRBIO Probe 1-Step Go No-ROX
qPCRBIO Probe 1-Step Go Separate-ROX
Ct 値がどれくらいの値を超えると結果は信頼できなくなりますか?
Ct 値はテンプレート濃度、反応の最適化、機器、研究室によって異なるため、カットオフ Ct 値を選択する際には注意が必要です。一般的に、35 を超える Ct 値は信頼できないと見なされ始めます。ただし、テンプレートのコピー数が少ない場合、反応が非効率的になると Ct が遅くなることがあります。相対的または絶対的な定量化方法を使用してカットオフを標準化することは常に良い方法です。また、遅く増幅された生成物を特定するために、生成物の融解曲線または電気泳動を実行して分析することをお勧めします。
qPCRBIO Probe 1-Step Go は 1 ステップ RT-PCR と 2 ステップ RT-PCR の両方に使用できますか?
いいえ、このキットは 2 ステップ反応には使用できません。2 ステップ反応の場合、より難しいターゲットには UltraScript® cDNA 合成キット または UltraScript® 2.0 cDNA 合成キット をお勧めします。また、ニーズに応じてリアルタイム PCR キットもお勧めします。
ROX は反応に悪影響を及ぼす可能性がありますか?
ROX (6-carboxy-X-rhodamine) は、主にウェル間の光路のばらつきによって生じる蛍光レベルの変動を正規化するために、ROX 依存型リアルタイム PCR 機器でパッシブ リファレンス 色素として使用されます。蛍光強度 (Rn) の正規化は、リアルタイム PCR ソフトウェアで、特定の信号の発光強度を ROX の発光強度で割ることによって行われます。
ROX は PCR 反応には関与せず、その蛍光レベルは各ウェルの DNA の量に比例しないため、この蛍光体を混合物に追加すると、増幅中に一定の蛍光信号が得られます。
パッシブ リファレンス スタンダードを必要とするさまざまなタイプのリアルタイム PCR 機器では、主に各システムの光学構成 (つまり、使用される励起源と光学系の種類) が異なるため、ROX の最適濃度が異なります。
ROX の添加量が少なすぎたり多すぎたりすると、非常にノイズの多い信号になり反応の結果に影響を及ぼすため、お客様にとって次のことが非常に重要です:
リアルタイム PCR の結果を最適化するために適切な ROX 濃度を決定する
反応を設定するために使用するソフトウェアで ROX 設定を確認する
最も一般的に使用されるシステム用の便利な選択ツール
HS Taq DNA ポリメラーゼの活性化時間を変更することはできますか?
ポリメラーゼを活性化するには、最低 2 分間使用することをお勧めします。酵素に悪影響を与えることなく、最長 15 分間まで長く使用することもできます。
1 ステップ反応と 2 ステップ反応のどちらが必要ですか?
1-Step
cDNA 合成と PCR 反応の両方が同じミックスで発生します。このオプションは、そのスピードとセットアップの容易さから、高スループット アプリケーションに適しています。汚染のリスクも軽減されます。低品質の RNA サンプルや、cDNA をアーカイブまたは別の分析に必要とする場合は、理想的ではありません。
2-Step
cDNA 合成と PCR 反応は別々に行われます。cDNA 産物を分析用に保持する必要がある場合は、このオプションの方が適しています。また、反応の最適化のレベルも高くなります。酵素の種類と濃度、RNA 入力、cDNA の濃度を制御できるため、1 ステップ形式に比べて感度が高くなります。
RT 反応で RNase 阻害剤を使用する必要はありますか?
いいえ、RTase Go には、劣化を防ぎ感度を高めるための RNase 阻害剤が含まれています。
生成物の低値または遅い Ct 値に関する一般的なトラブルシューティング
Ct 値が異常に遅い場合は、テンプレート RNA を希釈してみてください。これにより、存在する可能性のある阻害物質が反応を阻害しない濃度まで希釈されます。さらに、逆転写ステップを 55 °C に上げ、アニーリング/伸長温度を上げてみてください。これにより、RNA テンプレートやプライマーに存在する二次構造によって生じる問題の解決に役立つ場合があります。
反応阻害が関与している可能性がある場合は、テンプレートの量を減らすか[1]、反応に0.4~4.4 mg/mlのBSAを加えてみてください[2]。
より具体的な問題については、次の情報を添えてお問い合わせ
アンプリコンのサイズ
反応セットアップ
サイクリング条件
増幅トレースと融解プロファイルのスクリーンショット
Scipioni et al. A SYBR Green RT-PCR assay in single tube to detect human and bovine noroviruses and control for inhibition. Virology Journal.5:94 (2008). doi: 1186/1743-422X-5-94
Plante et al. The use of bovine serum albumin to improve the RT‐qPCR detection of foodborne viruses rinsed from vegetable surfaces. Applied Microbiology. 52:3 (2010) doi: https://doi.org/10.1111/j.1472-765X.2010.02989.x
qPCRBIO Probe Mix の蛍光が競合他社の製品で得られる蛍光と異なるのは正常ですか?
製品によって蛍光のプラトーが異なる場合があります。ただし、これは定量化の精度には影響せず、Ct 値は製品間で異なりません。
高感度 RT-PCR には mRNA の単離が必要ですか?
通常、mRNA の分離は必要ありません。qPCRBIO Probe 1-Step Go キットは、1 pg の総 RNA または 0.01 pg の mRNA を含むサンプルでも機能するように開発されています。
希少な mRNA 種を扱う場合は、感度を高めるために RT 反応で配列特異的プライマーを使用します。
プローブベースのミックスと色素ベースのミックスの違いは何ですか?
qPCRBIO Probe 1-Step Virus Detect などのプローブベースのキットは、感度が高く、非テンプレート増幅が現れる可能性は低くなります。マルチプレックスは、特定のプローブに対して異なる蛍光団を備えたアンプリコンを使用して測定できますが、色素を使用した場合はこれを実現できません。
qPCRBIO SyGreen 1-Step Detect や 1-Step Go などの色素ベースのシステムは、完全な dsDNA を検出するため、プライマー二量体やオフターゲット/非テンプレート増幅を表示します。これらは、融解曲線を分析することで、生成物のピークから分離できます。
ROX とは何ですか? それは必要ですか?
ROX はパッシブリファレンス色素であり、PCR 反応には関与しません。PCR に関連しない蛍光の変動を標準化するために使用されます。「リソース」セクションの qPCR 選択ツール
qPCRBIO Probe 1-Step Virus Detect ミックスの MgCl2 濃度はどれくらいですか??
すべての 4x qPCRBIO Probe 1-Step Go ミックスには、9 mM の濃度で MgCl2 が含まれています。つまり、反応の最終濃度は 4.5 mM です。
どのようなプライミング方法を使用できますか?
遺伝子特異的プライマーは 1 ステップ反応で使用できます。
マルチプレックス増幅を行う際に考慮すべきことは何ですか?
プライマーは、アニーリング温度が類似し、ターゲットに特異性があり、プライマー二量体を形成しないように設計する必要があります。プライマー/プローブ濃度は、発現レベルが大幅に異なる場合を除き、調整する必要はありません。発現レベルの高いターゲットの場合はプライマーの量を減らし、発現レベルが低いターゲットの場合はプライマーの量を増やす必要があります。
PCRBIO 1-Step Go RT-PCR キット、qPCRBIO Probe 1-Step Go、qPCRBIO SyGreen 1-Step Detect|Go キットの違いは何ですか?
PCRBIO 1-step Go RT-PCR キットは、エンドポイント RT-PCR 用に開発されました。qPCRBIO Probe 1-Step Go および qPCRBIO SyGreen 1-Step Detect|Go は、それぞれリアルタイム RT-PCR 用のプローブおよび色素ベースのオプションです。
マルチプレックスを行う場合、各プライマーの推奨濃度はどれくらいですか?
各プライマーは 0.4 µM の使用をお勧めします。この推奨濃度にはある程度の柔軟性がありますが、プライマー濃度をこの範囲を超えて増加させると酵素の活性に重大な影響を与える可能性があるため、増加させないでください。
このミックスはマイクロ RNA テンプレートに使用できますか?
はい、qPCRBIO Probe 1-Step Go はマイクロ RNA テンプレートに使用できます。専用のキットは販売していませんが、当社の RTase はすべて miRNA の定量化と分析に使用できます。
次の 2 つのアプローチのいずれかをお勧めします。
ユニバーサル RT プライマーを使用して、ポリ (A) またはポリ (U) テールを追加し (例: ポリ (U) ポリメラーゼによる)、続いてユニバーサルプライマーを使用して cDNA を合成します。[1], [2]
特定の RT プライマーを使用し、テーリング ステップ を省略します。[1],[3]-[5]
これらのアプローチの詳細がよくわからない場合は、ガイドラインとして以下のリストを参照してください。
Dave, V. P. et al. MicroRNA amplification and detection technologies: opportunities and challenges for point of care diagnostics. Lab Invest 99, 452-469, doi:10.1038/s41374-018-0143-3 (2019).
Mei, Q. et al. A facile and specific assay for quantifying microRNA by an optimized RT-qPCR approach. PLoS One 7, e46890, doi:10.1371/journal.pone.0046890 (2012).
Chen, C. et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res 33, e179, doi:10.1093/nar/gni178 (2005).
Raymond, C. K., Roberts, B. S., Garrett-Engele, P., Lim, L. P. & Johnson, J. M. Simple, quantitative primer-extension PCR assay for direct monitoring of microRNAs and short-interfering RNAs. RNA 11, 1737-1744, doi:10.1261/rna.2148705 (2005).
Androvic, P., Valihrach, L., Elling, J., Sjoback, R. & Kubista, M. Two-tailed RT-qPCR: a novel method for highly accurate miRNA quantification. Nucleic Acids Res 45, e144, doi:10.1093/nar/gkx588 (2017).