マルチプレックスプローブ 1 ステップ RT-qPCR を使用した SARS-CoV-2 および冬季ウイルスの検査パネル
Matteo Beretta PhD & Constantine Garagounis PhD
Introduction
冬季ウイルスは毎年繰り返される問題です。新型の SARS-CoV-2 ウイルスと相まって、診断が困難になっています。したがって、これらの病原体を確実に検出することが、適切な治療および保護隔離措置を適時に講じるために重要です。ウイルス株の識別におけるゴールドスタンダードは、全ウイルスゲノム配列決定または特定の株の主要なウイルス遺伝子のサンガー配列決定です。ただし、配列決定アプローチは時間がかかり、面倒です。したがって、RTqPCR による迅速なスクリーニングが推奨される方法であり、完全な診断に必要な場合は、その後に配列決定を行うことができます。RT-qPCR は、核酸ターゲットの検出と定量化のベンチマーク技術であり、非常に短時間で複数のサンプルとターゲットの信頼性の高い分析を可能にします。一般的な 40 サイクルの RT-qPCR 実行からの結果は、15 ~ 30 分のプレートセットアップ時間と、最新の機器によるほぼ即時の結果レポートと分析を考慮すると、3 時間未満で入手できます。
さまざまな冬季ウイルスを検出するために、いくつかの RT-qPCR プロトコルを使用できます。ただし、一般的な病原性ウイルス株を検出するためにマルチプレックス RT-qPCR を使用するのが最善のアプローチの 1 つです。これは、マルチプレックス化により、時間、試薬、機器を効率的に使用しながら、複数のターゲットを同時にテストできるためです。したがって、これは大容量の診断ワークフローに最適です。マルチプレックス RTqPCR は、検出するすべてのターゲットのプライマー ペアと特定のプローブをサンプルとともに 1 つの反応ミックスに組み合わせて実行されます。各プローブは個別の蛍光体でラベル付けされているため、サーモサイクリング中に固有の信号が記録されます。したがって、1 つのサンプルを 1 回の反応で複数のターゲットに対して検査できます。
このアプリケーションノートでは、一般的な冬季ウイルス(インフルエンザAウイルス、インフルエンザBウイルス、ヒトRSウイルスA2、および新型SARS-CoV-2ウイルス)を含む冬季パネルの、信頼性が高く迅速な(約1.5時間)マルチプレックスアッセイについて説明します。私たちは、現在のパンデミック後もこのような冬季パネルの使用が一般的になると考えており、これらの一般的な再発性病原体の診断テストを合理化できるようにこのアッセイを設計しました。
Method
PCR Biosystems 社では、SARSCoV-2 を含む冬季ウイルスパネルの検出プロトコルを確立しました。この目的のために、表 1 にリストされている、査読済みのジャーナルレポート 1 および WHO (世界保健機関/シャリテ・ベルリン プロトコル) と CDC (米国ジョージア州アトランタの疾病予防管理センター) のガイドライン 2 で診断の妥当性が特定および検証されたプライマーとプローブの配列を使用しました。また、このプロトコルでは、新しく開発した qPCRBIO Probe 1-Step Virus Detect RT-qPCR マスター ミックスを使用しました。テスト テンプレートは合成 RNA ウイルス配列で、各ウイルスのターゲット配列は、インフルエンザ A ウイルスのマトリックス タンパク質 M1 遺伝子、インフルエンザ B ウイルスのヘマグルチニン遺伝子、ヒト呼吸器合胞体ウイルス A2 のマトリックス タンパク質遺伝子、および SARS-CoV-2 ウイルスの E 遺伝子でした (表 1)。さまざまな条件を試験した後、これらのウイルスを検出するための最適化されたマルチプレックス RTqPCR プロトコルを紹介します。
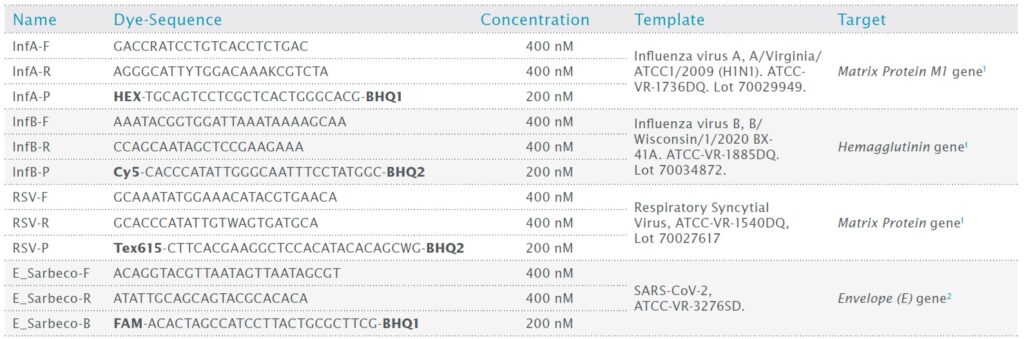
表1:使用したプライマー、プローブおよびターゲット
Primer and template preparation
各ターゲットについて、400μL 10μM プライマー (F + R) を 200μL 10μM プローブに混合してプライマー セットを調製しました (これは、0.8μL 10μM プライマー F + R と 0.4μL 10μM プローブを各 20μL 反応ミックスに加えることに相当します)。
テンプレートは、サンプルあたり 40,000 コピーから 4 コピーまで、5 ポイント、10 倍希釈系列で準備されました。各ターゲットにはテンプレート コントロール サンプル (NTC) は含まれていませんでした。各サンプルに対して 4 回の技術的反復を実行しました。
Practical Note:テンプレート ストックをチューブに一緒に追加して、各ウイルス ゲノムに対して 8000 コピー/μL の混合物を作成しました。
Reaction setup
反応は、Qiagen QIAgility ロボットを使用して、最終容量 20μL でセットアップされました。これには、4.2μL Milli-Q 水、5μL 4x qPCRBIO Probe 1-Step Virus Detect、1μL 20x UltraScript RTase、1.2μL x 4 プライマー/プローブ ミックス (上記参照)、および 5μL のプール RNA サンプルが含まれます。表 2 は、追加したコンポーネントの最終濃度をまとめたものです。
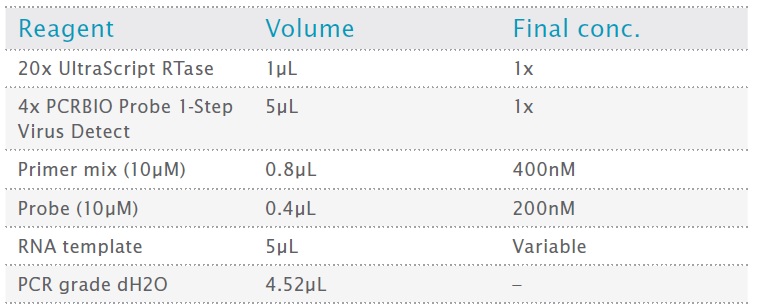
表2:反応のセットアップ
4 セットのプライマー ペア (F+R) を混合して、各オリゴヌクレオチドの最終濃度が 10 μM になるようにしました (例: 100 μM のプライマーの 8 つのストックのそれぞれに 10 μL、PCR グレードの dH2O 20 μL)。同様に、4 つのプローブを混合して、各最終濃度が 10 μM になるようにしました (例: 100 μM のプローブの 4 つのストックのそれぞれに 10 μL、PCR グレードの dH2O 60 μL)。
Cycling conditions
サーマルサイクリングは、表 3 に示すサイクリング パラメータを使用して、Bio-Rad CFX96 Touch™ qPCR マシンで実行されました。蛍光測定値は各サイクルで取得されました。

表3:サイクリング条件
Results
すべてのターゲットは、最低濃度(20μL反応あたり4コピー)でも40サイクル前に正常に検出され、NTCで生成物は検出されませんでした(図1および表4)。すべてのターゲットの効率は90〜110%で、R2値はすべてのケースで98%を超えました。すべてのターゲットの平均±SDターゲットCt値は、最高濃度(反応あたり40,000コピー)および最低濃度(反応あたり4コピー)でそれぞれ24.13 ± 1.02および37.30 ± 1.18でした。
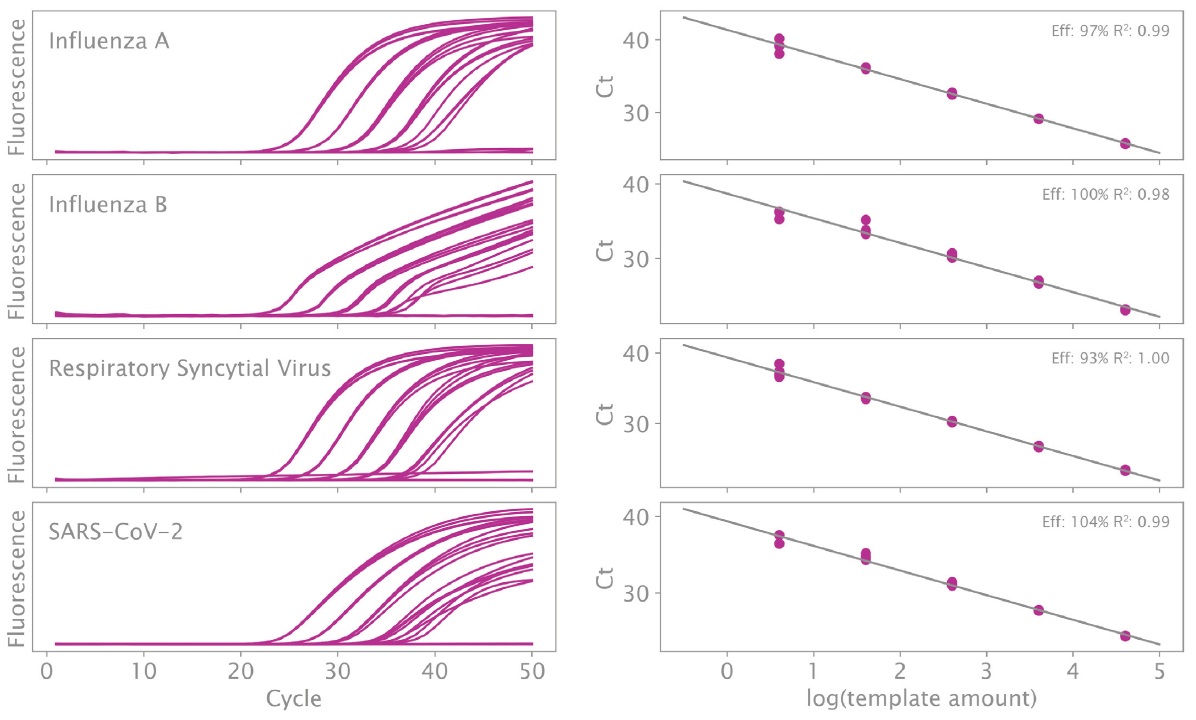
図1:インフルエンザA、インフルエンザB、RSウイルス、SARS-CoV-2ウイルスの4重検出
qPCRBIO Probe 1-Step Virus Detect を 4 回使用して、インフルエンザ A、インフルエンザ B、RS、および SARS-CoV-2 ウイルスの遺伝子を 4 重に増幅しました。増幅曲線は左側に、効率は右側に示されています。合成ウイルス RNA テンプレートの 5 つの連続希釈液が使用されました。これは、SARS-CoV-2 ゲノムの 40,000、4,000、400、40、および 4 コピーに相当します。総反応容量は 20 μL でした。InfA プローブは Hex で、InfB プローブは Cy5 で、RSV プローブは Texas Red で、SARS-CoV-2 E 遺伝子プローブは FAM でラベル付けされました。サイクル条件は、45°C で 10 分間の逆転写、95°C で 3 分間の変性、および 95°C で 15 秒、60°C で 30 秒の増幅を 50 サイクルでした。
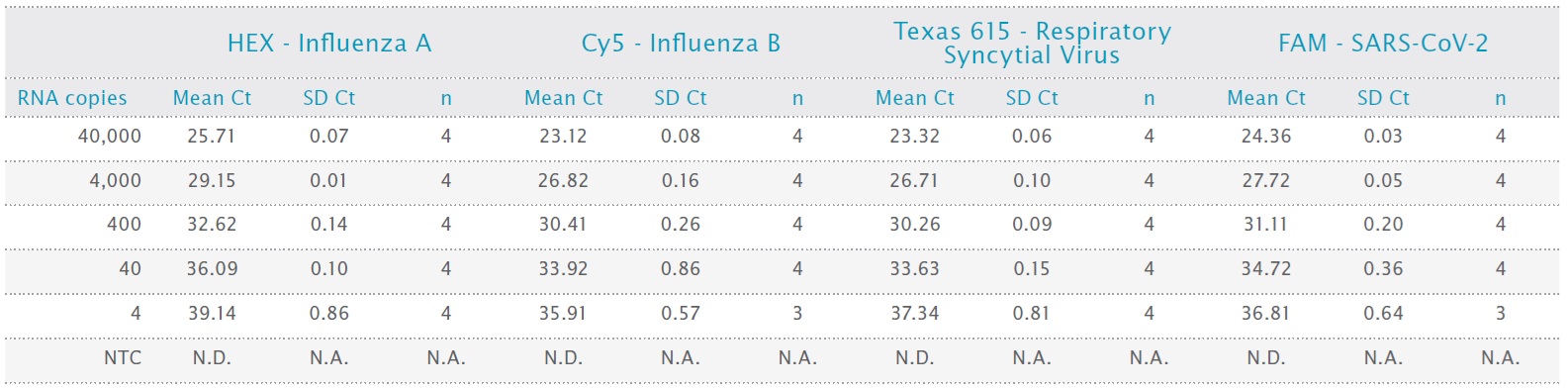
表4:図 1 に記載されている qRT-PCR のすべてのサンプルの平均 Ct 値と標準偏差値
Discussion
私たちのアプローチは、4 プレックス RTqPCR 反応 (20μL 反応あたり 4 コピー) で非常に少量のウイルス ターゲットを検出することに成功していることが証明されています。これにより、3 つの一般的な冬季ウイルスと新型 SARS-CoV-2 を 1 本の試験管で迅速かつ確実に検査できます。核酸ターゲットの陽性結果は必ずしも生存ウイルス粒子の存在を反映するわけではありませんが、RT-qPCR アプローチは依然として病原体の検出と定量化のベンチマーク メソッドであり、多くの臨床および研究診断手順の不可欠な部分となっています。これらの結果は、マルチプレックス RT-qPCR の威力と効率性も強調しており、この文脈では、このパネルに追加のターゲットを含めることができることを述べることが重要です。病原性ウイルス検出のための 10 プレックス RT-qPCR アッセイは成功しています [1]。さらに、特定の SARS-CoV-2 変異体用に設計されたプローブを含む他のターゲットを、現在のアプリケーションで選択したターゲットの代わりに使用できます。
本稿執筆時点では、COVID-19ワクチン接種やその他の予防策の導入が進んでいるにもかかわらず、SARS-CoV-2の懸念される変異体が絶えず出現しており、パンデミックからの脱出の障害となっています。そのため、他の一般的なウイルスに加えて、さまざまなコロナウイルス変異体の患者スクリーニングが重大な課題となっています。ECDC(欧州疾病予防管理センター)のガイドラインでは、SARS-CoV-2変異体の特定には、ウイルスゲノム全体の配列決定、または少なくともS遺伝子の完全なサンガー配列決定が必要とされています[3]。ただし、これらのアプローチは手間がかかり、時間がかかることから、RT-qPCRによるSARS-CoV-2のサンプルの事前スクリーニングも推奨されています。RT-qPCRは、配列決定が必要なサンプルの総数を減らし、コロナウイルス変異体の特定に対するより安価で迅速、高スループットの予備的アプローチを提供します。 RT-qPCRに加えて、いくつかの異なるアプローチを使用して特定の変異を事前にスクリーニングできますが、すべてのケースで、これらのアプローチの後にシーケンスを行って確認結果を得る必要があります[3]。
ECDCが予備的な変異体スクリーニングに推奨するRT-qPCRアプローチには、次のものが含まれます[3]。
- 複数の SARS-CoV-2 遺伝子をターゲットとするマルチプレックス RT-qPCR において、プライマー結合領域の変異または損失によって引き起こされる SARS-CoV-2 S 遺伝子のドロップアウトまたはターゲットの失敗
- 各ターゲット変異SNPごとに異なるプローブとコントロールを備えたプローブベースのRT-qPCR
適切なプライマーとプローブが選択されていれば、これらのアッセイは両方ともこのアプリケーション ノートに従って実行することができます。
PCR Biosystems 社の qPCRBIO Probe 1-Step Virus Detect は、新しいマルチプレックス アッセイの迅速なセットアップを可能にし、さまざまなテンプレート量、サンプル タイプ、ターゲット ヌクレオチド組成で信頼性が高くなるように設計されているため、面倒なマルチプレックス アッセイの最適化が不要になります。
Product use
qPCRBIO Probe 1-Step Virus Detect を含む PCR Biosystems 社製品は、単独では診断結果を提供せず、研究用途にのみ提供されます。ただし、すべての製品は ISO 13485 準拠の管理システムの下で製造されており、該当する国の法律で許可され、アッセイ自体の臨床検証が済んだ後、分子診断アッセイのコンポーネントとして使用するのに適しています。
どの製品がお客様のアプリケーションに最適かご相談されたい場合、または qPCRBIO Probe 1-Step Virus Detect を SARS-CoV-2 変異体検査に使用する方法についてさらに技術的なアドバイスが必要な場合は、お問い合わせください。
References
- Pretorius et al., Respiratory Viral Coinfections Identified by a 10-Plex Real-Time Reverse-Transcription Polymerase Chain Reaction Assay in Patients Hospitalized With Severe Acute Respiratory Illness—South Africa, 2009–2010, The Journal of Infectious Diseases, Volume 206, Issue suppl_1, 15 December 2012, Pages S159–S165.
https://academic.oup.com/jid/article/206/suppl_1/S159/984203 - Corman et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 2020 Jan; 25(3):2000045. doi: 10.2807/1560-7917.ES.2020.25.3.2000045. Erratum in: Euro Surveill. 2020 Apr; 25 (14): Erratum in: Euro Surveill. 2020 Jul; 25 (30): Erratum in: Euro Surveill. 2021 Feb;26(5): PMID: 31992387; PMCID: PMC6988269.
https://www.who.int/docs/default-source/coronaviruse/protocol-v2-1.pdf - European Centre for Disease Prevention and Control, World Health Organization. Methods for the detection and identification of SARS-CoV-2 variants. 3 March 2021. ECDC: Stockholm; 2021.
https://www.ecdc.europa.eu/en/publications-data/methodsdetection-and-identification-sars-cov-2-variants
